Schnellzugriffe
Meistgelesen
Schülerpraktikum GSH
- Text:
- Lars Pfeiffer
- Letzte Änderung:
- 05.05.2009
- Verantwortliche/r:
- Markus Thiemel
Schülerpraktikum GSH
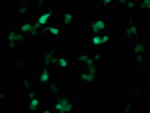
Während meines Praktikums beschäftige ich mich mit 3 verschiedenen Versuchsreihen deren Hauptthema die Apoptose, der programmierte Zelltod, war. Zuerst der Nachweis einer Genexpression von Säugerzellen durch DNA. Dies geschah mithilfe von 293T Zellen, einer speziellen Nierenzellkultur. Die Zellen wurden aus ihren Petrischalen mithilfe von Trypsin gelöst und nach Konzentrationsbestimmung in bestimmter Konzentration wieder ausgesät. Nach einer Nacht Inkubationszeit wurden die Zellen mit Plasmid-DNA transfiziert. Am nächsten Tag wurden Zelllysate hergestellt. Dazu wurden die 293T Zellen mithilfe von Ultraschall abgetötet und die Lysate nach mehrmaliger Zentrifugation und Aufreinigung entnommen. Die Proteinexpression wird über einen Western Blot nachgewiesen. Dazu werden die Zelllysate mit Probenpuffer vermischt und eine SDS-Gelelektrophorese wird durchgeführt. Danach wird der Blot angesetzt. Hierbei werden die Proteine vom SDS-Gel auf eine Membran übertragen. Diese Membran wird mit
Antikörpern behandelt, die an die Proteine anhaften. Durch einen zweiten, lumineszenzinduzierenden Antikörper wird eine ECL-Detektion durchgeführt. Prinzip dahinter ist die Detektion über Röntgenstrahlen, welche das von den Proteinen ausgehende Licht aufzeichnen.
Der zweite Teil bestand aus der Analyse der Genexpression von Wildtyp und einem speziell gezüchteten Maustyp.
Hierzu wurden B Zellen aus der Milz der entsprechenden Maus isoliert, stimuliert und daraufhin geerntet. Aus dem Zelllysat wurde aus mRNA mithilfe des Enzyms Reverse Transkriptase eine cDNA hergestellt, weil diese beständiger ist. Hierfür werden spezielle Primer benötigt, welche an die mRNA binden. Meist wird als Gegenstück zum Poly-A-Schwanz der mRNA ein Oligo-T-Abschnitt verwendet. Die DNA-Sequenzen lassen sich über PCR nun nachweisen und vermehren. Mittels einer quantitativen real-time PCR lässt sich die Expressionsrate der zugrundeliegenden mRNA darstellen.
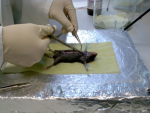
Die entstandenen DNA-Fragmente werden mittels Agarosegelelektrophorese aufgetrennt und können analysiert werden.
Der letzte Teil des Praktikums bestand aus der Proteinexpression in E.Coli Bakterien und darauf folgender Aufreinigung. Dazu werden E.Coli mit einem Plasmid, welches das Gen des zu exprimierenden Proteins trägt, transfiziert, und schließlich, nach einer Vermehrungsphase der Bakterien mittels IPTG (Isopropyl-Thiogalactosid) zur Proteinexpression angeregt.
Das Protein, das hergestellt werden soll, enthält üblicherweise einen Tag, eine als Markierung dienende bestimmte Basenabfolge, über die das Protein mithilfe von Antikörpern aufgereinigt werden kann.
Zur Aufreinigung werden die Bakterien im Ultraschall abgetötet und die Membranreste abzentrifugiert. Das Zelllysat wird mit der entsprechenden Sepharose inkubiert und mehrmals gewaschen. Dann wird das Protein wieder von der Sepharose gelöst und kann auf ein SDS-Gel aufgetragen werden.
 Follow @HvGG_edu
Follow @HvGG_edu